CURSO
GENÓMICA EVOLUTIVA Y POBLACIONAL
-2024 -
- Profesor: Dr Hernán Dopazo
- Docentes Auxiliares: Dra Francisca Almeida (JTP), Lic Johnma Rondon (Ayt I).
- Colaboradores: Bioing. Ezequiel Fernández, Dr. Jeremias Zubrzycki y Dr Pierre Luisi.
- Inscripción Oficial: 19/02/2024 al 03/03/2024
- Teóricas: Ma & Vi.17.00 a 19.00 hs. Comienza 19/03/2024. Martes, aula 116 entrepiso Pab 2.
- Trabajos Prácticos: Ma & Ju 10.00 a 13.00 hs. Comienza 21/03/2024. Laboratorio A. Primer Piso. Pab 2.
- Cuatrimestre: Primero
- Correlativas: TPs de Evolución aprobados.
- Modalidad: Clases teóricas, TPs en computadoras y exposición de seminarios. Ambos últimos obligatorios.
- Aprobación de TPs: 80% de asistencia. Parciales y recuperatorios aprobados con ≥ 5 puntos. Seminario grupal-oral aprobado. Recuperatorios luego del segundo parcial.
- Promoción: Aprobar los TPs con promedio de parciales ≥ 7 y final de Evolución aprobado.
- Final: Examen escrito u oral teórico-práctico y final de Evolución aprobado.
- Programa de Doctorado: 5 puntos, (120 hs). Ref Expte. Nº5700/2019
- Inscripción SIU Guaraní.
- Grado: Genómica Evolutiva y Poblacional (BIOL840152)
- Postgrado: Genómica Evolutiva y Aplicada (DOC8800294)
- Mail de contacto: genevopop_at_gmail.com
- Descarga el Programa Oficial Aprobado por CD (2019): Programa-CD-19-1870
Objetivos |
|
Que el alumno conozca los principales avances científicos y técnicos de la genómica, y se enfrente a los desafíos prácticos que impone el análisis masivo de estos datos en un marco de referencia evolutivo y poblacional.
|
|
mLas clases teóricas se organizan en cuatro módulos que analizan: 1- los adelantos en materia tecnológica y conceptual del estudio individual de los genomas humanos, y diversos tópicos de genómica comparativa y genómica funcional; 2- la diversidad genómica de las poblaciones humanas como fruto del desarrollo de consorcios internacionales para el estudio de la variabilidad en una especie modelo; 3- el uso de métodos estadísticos para descubrir los procesos que modelan la estructura del genoma, ya sea como consecuencia de sucesos ancestrales fortuitos, efectos demográficos frecuentes y eventos adaptativos únicos o convergentes, y 4- el estudio de la arquitectura genómica de caracteres simples y complejos, que determinan en el ámbito humano las particularidades que tiene la medicina de precisión, desde el estudio familiar de enfermedades mendelianas, hasta las perspectivas de modificar genomas utilizando herramientas de edición genética (1).
Las clases prácticas constan de 10 TPs cuyo común denominador es el manejo sistemático de datos genómicos en un sistema operativo abierto (Linux-Ubuntu), el uso de la consola y líneas de comando, programas estadísticos (R u otros de libre acceso), y scripts de Python. Los prácticos se realizan en una máquina virtual (virtualbox) para garantizar su funcionamiento en cualquier sistema operativo (dentro y fuera del curso). Las guías de TPs son accesibles desde un entorno Git (2). El 90% de los TPs utilizan datos de NGS humanos. Los seminarios son preparados por los alumnos, quienes de forma grupal o individual, presentan un tema de actualidad en un área de interés explicando hipótesis, métodos y evaluando críticamente los resultados y conclusiones. ________________ (1) El énfasis en temas humanos es una excusa para extrapolar el enorme potencial de aplicaciones genómicas hacia otros modelos biológicos. (2) No es necesario tener conocimientos previos de estos lenguajes para realizar el curso. |
Teóricas |
|
Tema 1: genomas Humanos y tecnologías de secuenciación
El proyecto genoma humano. Genoma diploide. Resecuenciación de genomas humanos. Tecnologías de secuenciación: primera, segunda y tercera generación: desde la tecnología Sanger a los Nanoporos. Formación de librerías de NGS (Next Generation Sequencing). El genoma de referencia y la representación del pan-genoma. 'Big data' genómico y computación en la nube.
Tema 2: genómica funcional
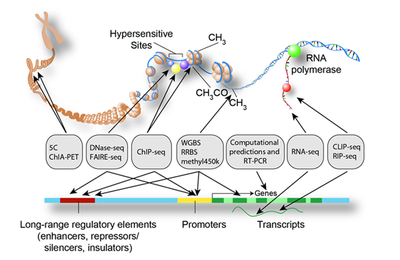
Enciclopedia de los elementos regulatorios del genoma: ENCODE y proyectos similares. Marcas epigenéticas de la expresión génica. Mapa del epigenoma humano. Tecnologías para la expresión masiva de genes: Tiling-arrays, DNase-seq, FAIRE-seq, Chip-seq, RNA-seq. Elementos ultraconservados del genoma humano. Críticas evolutivas a la visión funcional extendida de los genomas eucariotas. La definición de gen en la era postgenómica.
Tema 3: genómica comparativa
El origen de la arquitectura genómica. Complejidad y tamaño efectivo de las poblaciones. Teoría de peligrosidad mutacional en genomas. Modelos de neo y sub- funcionalización de genes duplicados. Modelos neutros de ecología en genomas eucariotas. Principales transiciones evolutivas. Origen de la individualidad y los niveles de selección. Cooperación y conflicto.
Tema 4: variabilidad genómica poblacionAL
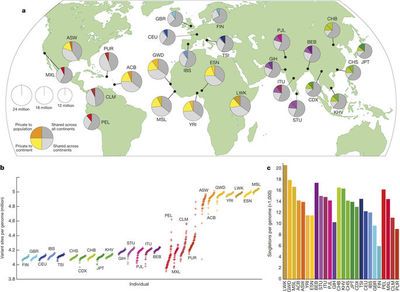
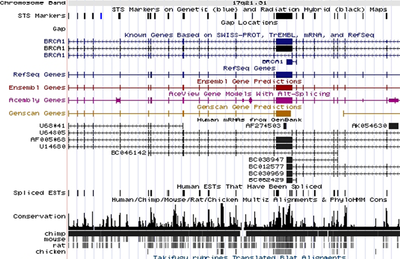
Polimorfismos de un único nucleótido (SNP's). Tag-SNPs. Haplotipos y bloques de haplotipos en el genoma humano. Proyecto HapMap y 1.000 Genomas Humanos. Imputación de genotipos. Mecanismos de formación de variantes en el número de copias (CNVs). Variación estructural en el genoma humano. Mapeo de variantes estructurales a partir de datos de NGS. Comparación de la variabilidad genómica en otros eucariotas.
Tema 5: demografía y estructura poblacional
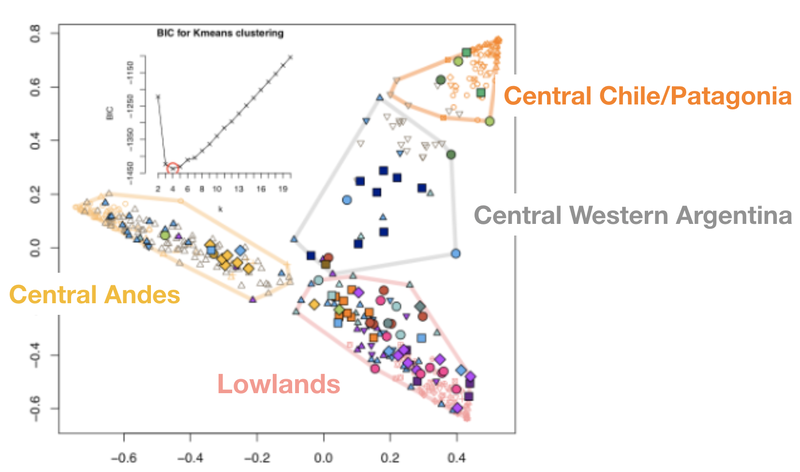
Secuenciación en profundidad de regiones genómicas. Espectro de frecuencias a partir del alelo menor (MAF) y el derivado. Modelos demográficos en poblaciones. Métodos para la caracterización de la historia evolutiva de las poblaciones: "PCA, Structure, Treemix, Hapmix". Estructura genómica de las poblaciones humanas. Comparación con otras especies eucariotas.
Tema 6: poblamiento continental Y GENOMAS ANCESTRALES
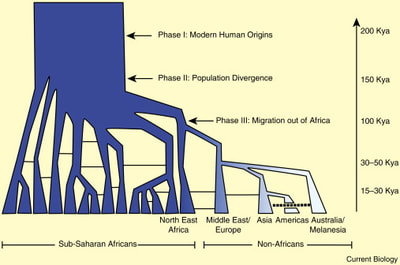
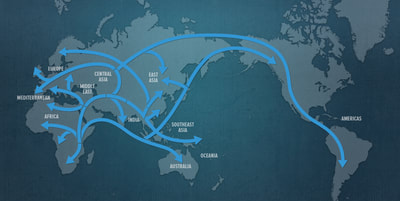
Evidencias del modelo serial de efecto fundador de la salida de Africa de los humanos modernos. Evidencias genómicas del origen sudafricano de los humanos modernos. Genoma de Homínidos Ancestrales: Neanderthals y Denisovanos. Evidencias de hibridación humana con otros genomas de homínidos. Poblamiento del continente americano. Concepto de raza, etnicidad y ancestría. Interacción entre genomas y cultura.
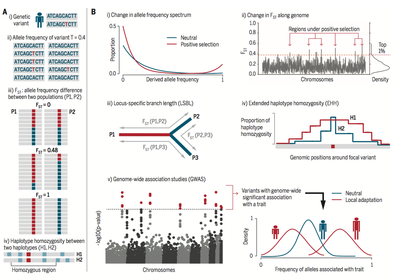
Tema 7: Pruebas de neutralidad y selección Natural en genomas completos
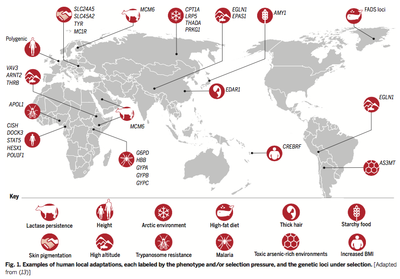
Estadísticos de selección natural en secuencias codificantes y no-codificantes del genoma: Tajima, Fay y Wu, McDonald-Kreitman, dN/dS y test del largo de los haplotipos. Técnicas de “sliding-windows”. El rango temporal de aplicación de estadísticos en el genoma humano. El mapa de la adaptación genómica ancestral y reciente en el genoma humano. Estudios de selección en otros genomas eucariotas. Selección genómica. Aplicaciones en ganadería y agricultura.
Tema 8: Genómica de enfermedades
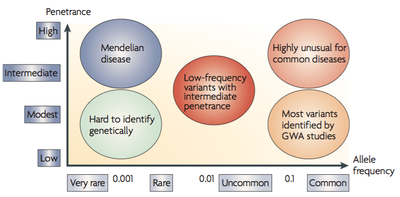
La arquitectura genética de la enfermedades. El interactoma y la visión de la red de enfermedades. Estudios de asociación de genoma completo. Replicabilidad. Hipótesis de enfermedades comunes / variantes comunes. El enigma de la heredabilidad oculta. El proyecto ExAC y GnomAD. Exomas y su aplicación en enfermedades raras. Pleiotropía antagonista y senescencia humana.
Tema 9: medicina DE PRECISión I
Diagnóstico genético dirigido al consumidor. Genómica reproductiva. Pruebas de enfermedades monogénicas en heterocigosis. Diagnóstico genético preimplantacional de aneuploidías y enfermedades. Karyomapping. Pruebas genómicas prenatales y postnatales. “Cell-free” DNA y estudios no-invasivos del embrión. Paneles de genes para enfermedades. El consorsio UK Biobank.
Tema 10: medicina DE PRECISIÓN II
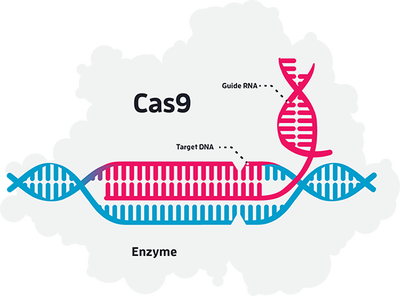
Genómica del cáncer. “Tumor-free” DNA. El problema de la heterogeneidad genética de los tumores. El cáncer como un problem evolutivo. Genómica de Células Únicas. Microbiomas humanos y ecológicos. Edición genética con Crispr-Cas9.
TRABAJOS Prácticos
TP1- explorando datos genómicos en la WEB
Exploradores web de datos genómicos. Estructura y uso del Ensembl. Búsqueda de variantes del genoma. Acceso y extracción de datos del genoma humano usando BioMart.
TP2- INtroducción al Sistema Operativo Linux
La consola de Linux. Los comandos básicos de la consola. Resumen sobre el sistema de ficheros de Linux. Permisos de usuarios y de grupos. "Scripting" básico. Corriendo blast local como ejemplo de uso.
TP3- control de calidad y preprocesamiento de DATOS NGS
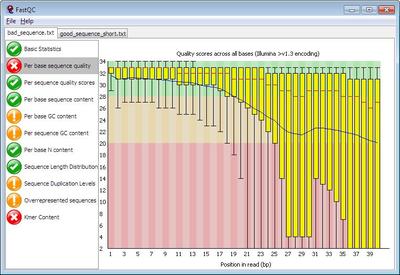
Control de calidad de las lecturas de NGS utilizando FastQC. Procesamiento de las lecturas de los datos NGS utilizando FASTX-toolkit y cutadapt.
TP4- Introducción al ensamblado genómico
Introducción al ensamblado de los datos de NGS (estrategias, métodos, errores frecuentes, espectro "k-mer", y gráficos de de Bruijn). Ensamblado con ABYSS. Estimación de parámetros y ensamblado con VELVET. Alineamiento al genoma de referencia utilizando BOWTIE2.
TP5- RNAseq y ANÁLISIS DE LA EXPRESIÓN GÉNICA DIFERENCIAL
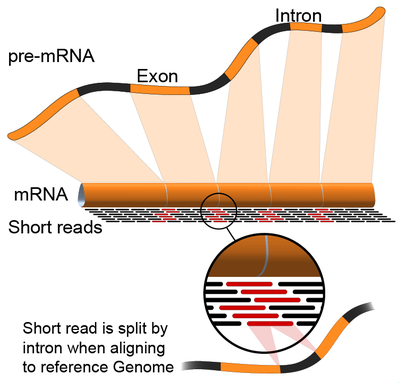
Alineamiento de lecturas de RNA-Seq a una referencia genómica. Conteo de lecturas y calculo de expresión génica. Análisis de expresión diferencial. Exploración de datos con explorar las webs de g:Profiler, HumanMine, Reactome y STRING.
TP6- ANALISIS DE VARIANTES ESTRUCTURALES
Análisis de Variantes Estructurales con NGS. Comparación de variantes entre bases de datos. Acceso a la Base de Datos de Variantes Genómicas. Predicción de Variantes Estructurales y Visualización utilizando inGAP-sv.
TP7- ANÁLISIS DE HAPLOTIPOS Y VARIABILIDAD GENÓMICA POBLACIONAL
"Variant Call File format (VCF)". Extracción y análisis de datos de 1,000 genomas con tabix y VCF tools. Desequilibrio de ligamiento, Tagging SNP’s, Haplotipos en Fase. Manipulación de los datos utilizando Haploview.
TP8- ANÁLISIS DE LA ESTRUCTURA POBLACIONAL
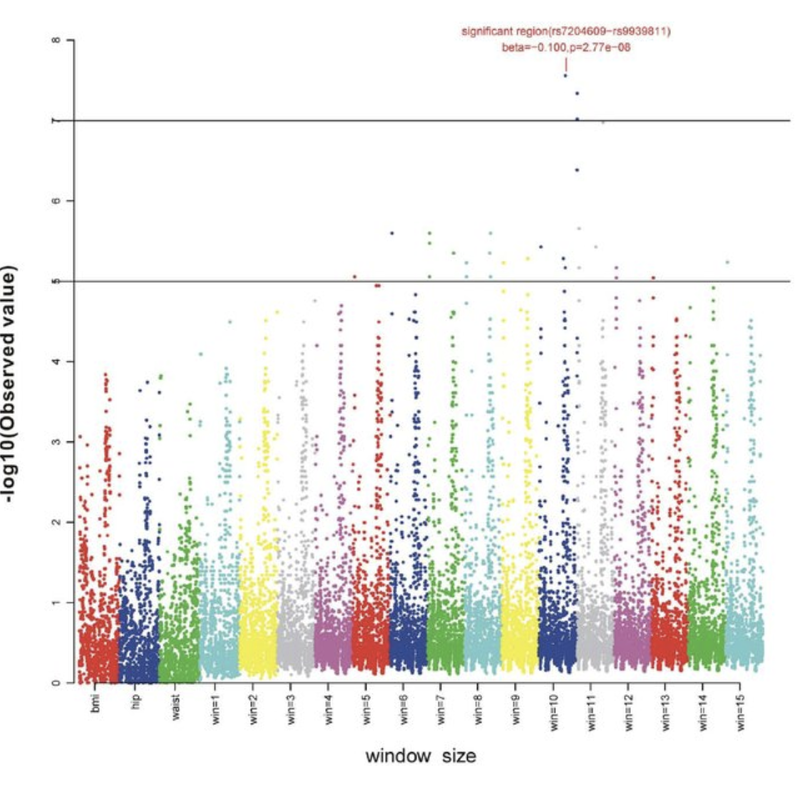
Filtrado de SNPs en desequilibrio de ligamiento. Computación y representación del espectro de frecuencias del alelo mínimo (MAF). Análisis de componentes principales con datos genómicos (PCA). Manejo de datos genotípicos con plink. Análisis de ancestría global con Admixture. Reconstrucción de haplotipos desde los genotipos con shapeIT. Inferencia de ancestría local con RFMix. Análisis en componentes principales de ancestría específica (ASPCA) con smartpca. Imputación de genotipos con impute2. Asociaciones falsas por estructuración de datos. Manhattan Plots. Q-Q Plots.
TP9- ANÁLISIS DE SELECCIÓN NATURAL EN EL GENOMA HUMANO
Análisis estadístico de la adaptación genómica utilizando DNAsp. Pruebas de SN con PBS, iHS en el genoma humano. Análisis de adaptación utilizando el browser de selección de 1,000 genomas.
TP10- GWAS. ANOTACIÓN DE VARIANTES GENÉTICAS
GWAS utilizando PLINK. GATK Best Practices Pipeline: Preprocesamiento, re-alineamiento local alrededor de indels, recalibración del score de calidad por base, descubrimiento de variantes.
Libros recomendados
Si bien ningún libro cubre completamente todo el programa del curso, a continuación listamos algunos que suelen ser útiles y didácticos.
- Michael Lynch (2007). The Origins of Genome Architecture. 1st edition. Link
- Jobling M., Hollox E., Kivisild T., Tyler Smith C. (2014). Human Evolutionary Genetics. 2nd edition. Link
- Dan Graur (2016). Molecular and Genome Evolution. 1st edition. Link
- Carlberg C., & Molnár F. (2016). Mechanisms of Gene Regulation. 2nd edition. Link
- Balding, D., Moltke, I. & Marioni, J. (2019). Handbook of Statistical Genomics 4 ed. eds, Wiley. Link
Calendario de Teóricas y Prácticas